
 Retour à l'accueil
Retour à l'accueil
Le laboratoire
Des capacités reposant sur des plateaux techniques dédiés à vos projets, et des partenaires de qualité
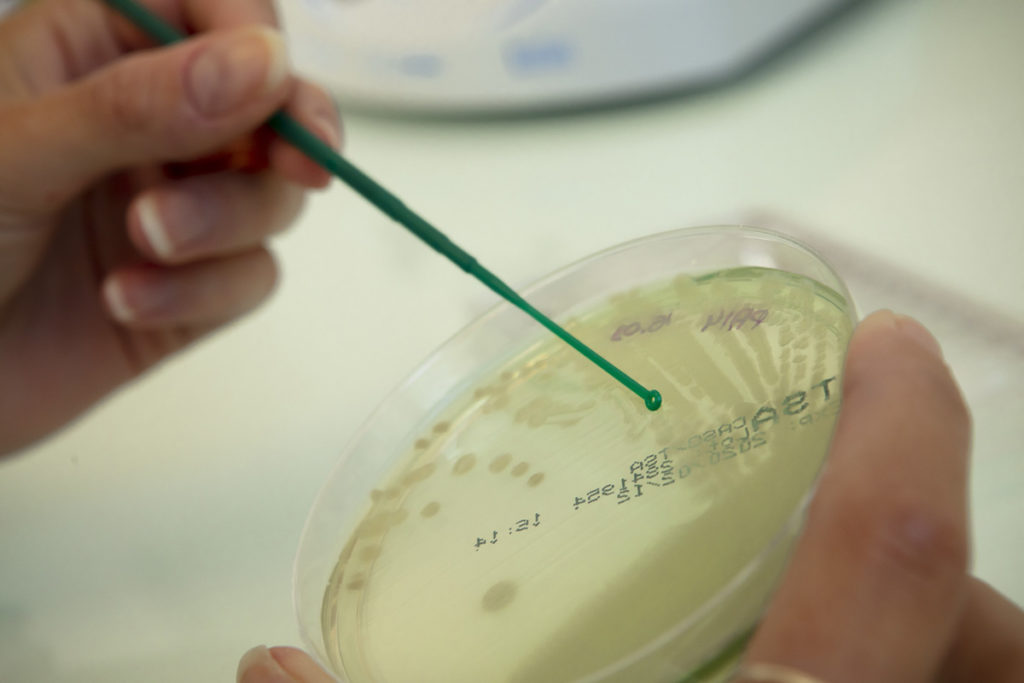
MICROBIOLOGIE
Notre plateforme de microbiologie nous permet de manipuler des micro-organismes de nature diverse : bactéries, levures, moisissures, phages, dont un grand nombre de souches pathogènes. De plus, l’acquisition d’une station anaérobie est un élément important qui nous permet aujourd’hui de cultiver des souches qui peuvent s’avérer exigeantes en termes de conditions atmosphériques de croissance.
Le plateau de microbiologie en bref, c’est :
- 125 m² de laboratoire respectant le niveau 2 de sécurité biologique (BSL2)
- Une station anaérobie
- Plusieurs systèmes analytiques tels que des lecteurs de microplaques Tecan Spark 10M ou BMG Labtech Spectrostar Nano
- Des systèmes de comptage de micro-organismes (Bactobox et Spiral platter Eddy Jet)
- Des électroporateurs
- Une ultracentrifugeuse
- Des enceintes climatiques thermo-contrôlées
- Une plateforme de spectrométrie de masse Maldi-Tof de Bruker® déportée
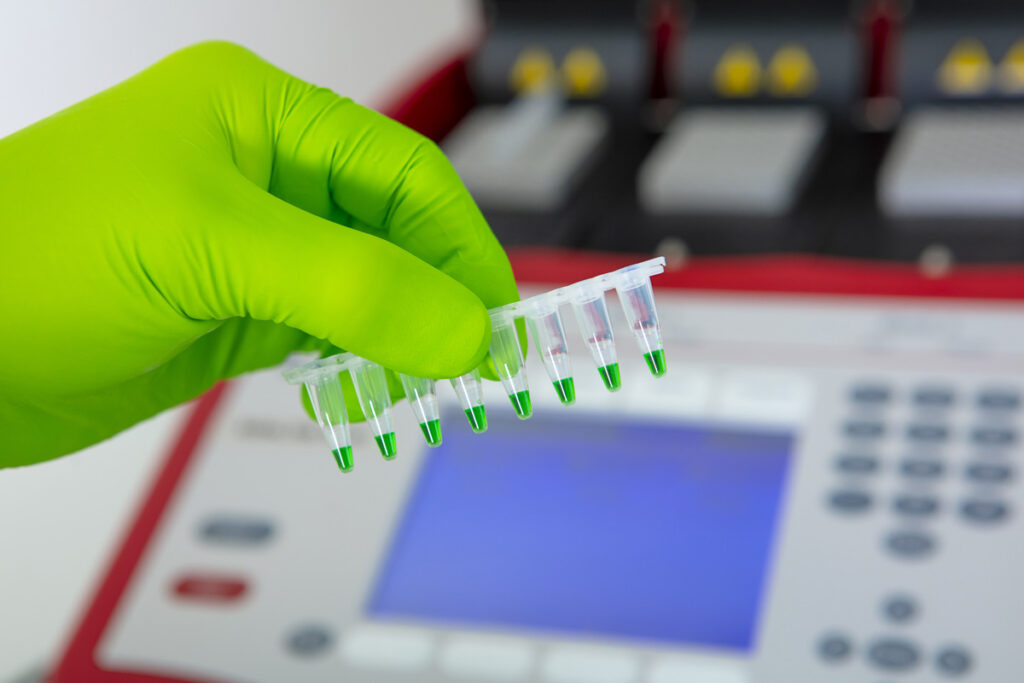
BIOLOGIE MOLECULAIRE
Notre plateforme de biologie moléculaire nous permet de remplir des missions variées telles que l’ingénierie génétique ou l’étude d’expression génique. Ce plateau technique dédié à la biologie moléculaire se combine à la plateforme de microbiologie, les projets nécessitant souvent l’emploi des deux compétences.
Le plateau de biologie moléculaire en bref, c’est :
- 46 m² de laboratoire respectant le niveau 2 de sécurité biologique (BSL2)
- Des Thermocycleurs Biometra Trio 48
- Des systèmes de PCR en temps réel
- Des systèmes d’électrophorèse et de visualisation d’ADN
- Des systèmes de quantification d’acides nucléiques
- Des enceintes climatiques thermo-contrôlées

BIOLOGIE CELLULAIRE
Notre plateforme de biologie cellulaire nous permet de manipuler différentes lignées cellulaires eucaryotes et de les mettre au contact de micro-organismes. La manipulation de ces cellules nous permet de proposer des services d’évaluation de cytotoxicité et de viabilité cellulaire, en présence de composés, de bactéries, de micro-organismes fongiques ou de phages. De plus, ces modèles nous permettent d’étudier de façon précoce des paramètres d’infection. Le recours à des lignées cellulaires eucaryotes s’avère également utile pour l’étude ou la production de bactéries intracellulaires.
Le plateau de biologie cellulaire en bref, c’est :
- 32 m² de laboratoire respectant le niveau 2 de sécurité biologique (BSL2)
- Un microscope à fluorescence inversé
- Des banques cellulaires stockées dans l’azote liquide – sur une plateforme déportée
- Un lecteur Tecan Spark 10M pour le comptage cellulaire automatisé
Notre exigence qualité
Ces plateaux techniques bénéficient d’équipements variés de haute technologie, validés par les équipes du laboratoire, nous permettant de réaliser des prestations de qualité. Smaltis fait d’ailleurs régulièrement l’acquisition d’appareils pour répondre à de nouveaux projets et rester à la pointe de la technologie.
Smaltis travaille avec des fournisseurs de matériel, de réactifs, et d’équipements qualifiés, avec lesquels une relation de confiance est établie, pour certains depuis les débuts de l’entreprise. Nous avons à cœur d’utiliser du matériel de qualité et robuste, essentiel au bon déroulé des manipulations et à la fiabilité des résultats obtenus. Nous sélectionnons ainsi le meilleur, pour vous donner le meilleur !
Les portes de notre laboratoire sont toujours ouvertes, n’hésitez à venir visiter nos locaux et nos plateformes !
Un réseau étendu de partenaires et de groupes de travail
Afin d’apporter des solutions clé en main, Smaltis s’est entourée de nombreux partenaires. Smaltis est également membre de différents réseaux pour rester au contact de nombreux acteurs dans le domaine de la microbiologie appliquée à la santé, et de bien cerner les besoins du secteur.
Rejoindre POLEPHARMA, c’est intégrer un territoire de mutualisation des besoins, des savoir-faire et des solutions où chacun contribue à l’excellence de tous. Rejoindre POLEPHARMA, c’est prendre part au réseau économique et industriel des acteurs de la première filière pharmaceutique française : Laboratoires de recherche, Sites de production, Façonniers, Fournisseurs, Centres de formation.
En savoir plus
L’AFSSI, l’Association Française des Sociétés de Services et d’Innovation pour les Sciences de la Vie est née en 2012 de la volonté de fédérer les sociétés françaises de services et d’innovation technologique dans le domaine stratégique des Sciences du Vivant. L’AFSSI vise à regrouper tous les secteurs de la biotechnologie, chimie, environnement, cosmétologie, agroalimentaire, bioinformatique, et inclut le diagnostic et les essais cliniques.
En savoir plus
Labellisé pôle de compétitivité à sa création, Lyonbiopôle Auvergne-Rhône-Alpes est le catalyseur de l’écosystème d’innovation santé en Auvergne-Rhône-Alpes, dont il assure la connexion, le développement et la promotion.
En savoir plus
MabDesign, l’association française de la filière industrielle des biomédicaments. Créé en 2014 en réponse à l’Appel à projet «Filière Industriel Stratégique d’Avenir», et en réponse à une proposition du précédent CSF Bioproduction, MabDesign vise à structurer la filière industrielle des biomédicaments en France depuis ses phases de R&D jusqu’à la mise sur le marché, en passant par la bioproduction et à générer la création de start-up innovantes issues de la recherche académique française.
En savoir plus
Pôle de compétitivité labellisé par l’Etat depuis 2005, le PMT est constitué en Association Loi 1901.
Notre vocation : catalyser l’innovation et accélérer le business des entreprises industrielles en Bourgogne-Franche-Comté. Nos actions ? Accompagner au quotidien et de manière individualisée plus de 240 adhérents industriels issus de la sous-traitance de spécialité ou développant des produits à forte valeur ajoutée. Les accompagnements proposés découlent des structures qui nous financent. Pour en savoir plus, découvrez nos différents programmes.
En savoir plus
Un groupe basé sur la complémentarité d'expertise avec un savoir-faire pluridisciplinaire. Une gamme de solutions intégrées en biotechnologie. Une plateforme complète, de l’ADN à l’anticorps recombinant. Services sur mesure et produits en Biotechnologie : Biologie Moléculaire – Immunologie – Ingénierie des protéines et des cellules. Une synergie efficace au sein d’une équipe d’experts.
En savoir plus

Une implantation stratégique
Smaltis s’est implantée, dès sa création, en plein cœur de la zone TEMIS Santé à Besançon, construite autour du Centre Hospitalo-Universitaire Jean-Minjoz, de l’Etablissement Français du Sang et de l’INSERM. Ce parc d’activités a pour objectif de devenir un pôle national du génie biomédical. C’est en toute logique que Smaltis s’est installée au cœur de ce réseau dynamique, afin d’être un véritable acteur de cette aventure et de cet objectif, tout en étant entourée de partenaires tournés vers la recherche et l’innovation en santé, tels que RD-Biotech, Skinexigence, Macopharma, Lymphobank, ISIFC et Biotika Biomedical.
Contactez-nous




















